`Rdkit & OpenBabel Tutorial
本教程主要参考了Mr. Chen Blog (main source)、Rdkit官方文档和个人使用经验(多种文件转化为CM描述符程序使用指南)
Chapter 1- 为Mr. Chen Blog的转载
Chapter 1 Introduction
RDKit是什么?
开源的化学信息工具包
- 采用了商业友好的BSD协议
- 核心数据结构和算法用C++实现
- 通过Boost.Python技术对RDKit进行封装,提供Python2/Python3的接口
- 通过SWIG技术提供Java 和C# 接口
- 提供了大量对化学分子2D/3D的计算操作
- 生成用于机器学习的分子描述符
- 基于PostgreSQL搭建分子数据库
- KNIME中的化学信息计算支持(https://www.knime.com/rdkit)
RDKit其他信息
RDKit官网: http://www.rdkit.org
支持多种系统: Mac/Windows/Linux
版本发布周期: 6个月
Web上的资源
- 官网: http://www.rdkit.org
- Github:(https://github.com/rdkit)
- Sourceforge (http://sourceforge.net/projects/rdkit)
- 博客: (https://rdkit.blogspot.com)
- 教程: (https://github.com/rdkit/rdkit-tutorials)
- KNIME和RDKit的集成 (https://github.com/rdkit/knime-rdkit)
社交媒体:
- Twitter: @RDKit_org
- LinkedIn: https://www.linkedin.com/groups/8192558
- Slack: https://rdkit.slack.com(需要加入的话,联系Greg)
教程:
“Getting Started width the RDKit in Python”,Greg Landrum’s RDkit blog,and a particularly nice example from the ChEMBL-og.
发展过程
- 2000-2006: 为了建立ADMET、生物活性等性质的预测模型,开发了相应的工具。
- June 2006: 发布该工具(BSD license)
- 至今: 诺华内部支持继续更新和完善该软件,并对社区开源。
和其他开源工具整合
- KNIME:工作流程和分析工具
- PostgreSQL: 可拓展关系数据库
- Django:python的web框架 :“締め切りに追われる完璧主義者のためのウェブフレームワーク”
- SQLite:使用最多的数据库,最简单的数据库
- Lucene :文本搜索引擎[#f1]_
使用RDKit的开源工具
- `Open Force Field Toolkit https://github.com/openforcefield/openforcefield/’__ - 于直接化学感知的力场参数引擎
- stk - 自动设计分子的python包
- gpusimilarity - 基于gpu的分子指纹搜索工具
- Samson Connect - 纳米系统建模仿真软件
- mol_frame - 为Dask和Pandas 提供处理化学结构的接口
- RDKitjs - JavasScript封装了RDKit的部分功能
- DeepChem - 用于化学领域深度学习的python包
- mmpdb - Matched molecular pair database generation and analysis
- CheTo - Chemical topic modeling
- OCEAN - Optimized cross reactivity estimation
- ChEMBL Beaker - RDKit 和 OSRA 的独立 web 服务
- myChEMBL - A virtual machine implementation of open data and cheminformatics tools
- ZINC - 用于虚拟筛选的可购买化合物开源数据库
- sdf_viewer.py - sdf 查看器
- sdf2ppt - 把sdf转换成PPT
- MolGears - A cheminformatics tool for bioactive molecules
- PYPL - Simple cartridge that lets you call Python scripts from Oracle PL/SQL.
- shape-it-rdkit - Gaussian molecular overlap code shape-it (from silicos it) ported to RDKit backend
- WONKA - 蛋白配体复合物结构分析和查询的服务
- OOMMPPAA - Tool for directed synthesis and data analysis based on protein-ligand crystal structures
- OCEAN - web-tool for target-prediction of chemical structures which uses ChEMBL as datasource
- chemfp - 快速分子指纹相似性搜索
- rdkit_ipynb_tools - 用于jupyter notebook的rdkit工具
- Vernalis KNIME nodes
- Erlwood KNIME nodes
- AZOrange
- Contrib目录
contrib目录位置: https://github.com/rdkit/rdkit/tree/master/Contrib 。 contrib目录,是RDKit发布版本的一部分,包含社区成员贡献的代码。
Chapter 2 Installation
anaconda安装方式适合不同的操作系统(强烈推荐)。
anaconda python
anaconda介绍
anaconda python 是非常好用的python解释器,自带多种好用功能,如:建立python虚拟环境、conda软件包管理工具等。 conda 命令包能够非常方便的安装各种预编译好的软件包,常用的软件通常都为提供相应的conda软件包。 conda 命令也可以帮忙创建独立的python环境。
安装anaconda python
去anaconda 官网 <https://www.anaconda.com/distribution/#download-section>__ 根据操作系统,下载相应的anaconda python 软件包安装即可。 推荐anaconda 3,自带python3 解释器。
windows 先安装rdkit
我的电脑是win10,64位。以我的电脑为例,首先下载 anaconda python 3.
下载anacond pyhon
注解
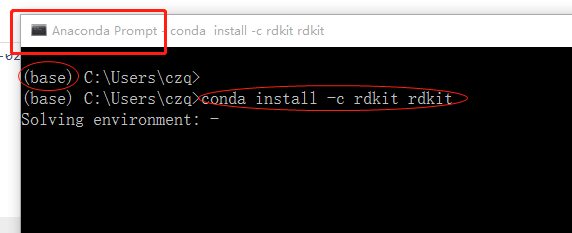
打开 anaconda promt.
这里我就直接在base环境下安装rdkit,
xxxxxxxxxxconda install -c rdkit rdkit
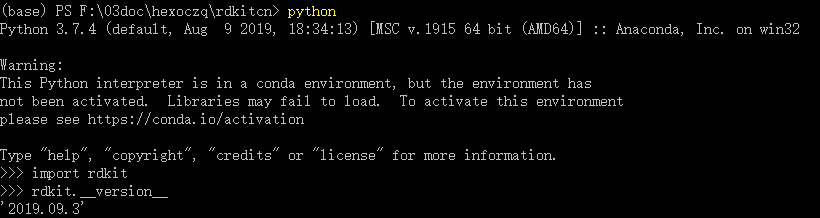
在anaconda promt 中测试是否安装成功。在cmd中输入
xxxxxxxxxxpython>import rdkit>rdkit.__version__
如果能看到rdkit的版本则说明安装成功, 如下图所示。
linux下安装rdkit
- step1 去
anaconda 官网 <https://www.anaconda.com/distribution/#download-section>__ 下载linux版本的Anaconda3*.sh文件;
- step2 在终端中运行
xxxxxxxxxxsh Anaconda*.sh
- step3 用conda命令安装rdkit
conda install -c rdkit rdkit
conda的详细使用参见python programming模块
Chapter 3 基础教程
[Getting Started with RDKit in Python]
基础教程主要对RDKit有一个初步的了解,明白RDKit能做什么,一些常用的功能。 RDKit功能的算法细节,参见高级教程或者参考文献。
注解:
- 推荐使用Jupyter Notebook 工具,学习RDKit.
- 从2019.03版本及其后续版本不再支持python 2, 支持python2的最后一版是 2018.09。
- rdkit 的版本号 从 1版开始,启用年月的版本号。半年发布一次版本。
现在python 3已经很成熟了,python 2 有的包 python 3 也有了,让我们一起拥抱python 3吧。
本教程中使用到的素材有:
- 2D分子: input.mol
- 2D分子sdf: 5ht2ligs.sdf
- 3D分子: 1t3r_lig.mol
1. 读写分子操作
读分子操作:
RDKit 支持从Smiles、mol、sdf 文件中读入分子获得分子对象。 |Smiles、mol 是通常用于保存单个分子;而sdf格式当初是作为分子库形式设计的。 因此读入sdf得到的是分子迭代器,读入Smiles 和mol 文件是分子对象。
示例1: 读入smiles
x
smi='CC(C)OC(=O)C(C)NP(=O)(OCC1C(C(C(O1)N2C=CC(=O)NC2=O)(C)F)O)OC3=CC=CC=C3'from rdkit import Chemfrom rdkit.Chem import AllChemmol = Chem.MolFromSmiles(smi)print(type(mol))显示mol的类型: rdkit.Chem.rdchem.Mol
示例2: 读入mol 文件
from rdkit import Chemmol = Chem.MolFromMolFile('rd.mol')print(type(mol))显示mol的类型: rdkit.Chem.rdchem.Mol
示例3: 读入sdf文件
from rdkit import Chemmols_suppl = Chem.SDMolSupplier('2d.sdf')print(type(mols_suppl))|显示变量mols_suppl的类型:<class ‘rdkit.Chem.rdmolfiles.SDMolSupplier’>。 |mols_suppl 可以看成是mol的列表,支持索引操作和迭代操作。
mol_1= mols_suppl[0]print(type(mol_1))for mol in mols_suppl:print(type(mol))写分子操作:
RDKit 可以把分子对象保存成Smiles、molBlock、mol文件。
示例:
xxxxxxxxxxsmi='CC(C)OC(=O)C(C)NP(=O)(OCC1C(C(C(O1)N2C=CC(=O)NC2=O)(C)F)O)OC3=CC=CC=C3'mol = Chem.MolFromSmiles(smi)smi = Chem.MolToSmiles(mol)print(smi)molblock = Chem.MolToMolBlock(mol)print(molblock)print(molblock,file=open('foo.mol','w+'))print(*objects, sep=’ ‘, end=’n’, file=sys.stdout, flush=False) python的print函数file参数支持定义输出位置。
2. 操作分子
获取分子中的原子
GetAtoms()mol对象中有获取所有原子的方法
GetAtoms(); - mol.GetAtoms()示例:
xxxxxxxxxxfrom rdkit import Chemsmi='CC(C)OC(=O)C(C)NP(=O)(OCC1C(C(C(O1)N2C=CC(=O)NC2=O)(C)F)O)OC3=CC=CC=C3'mol = Chem.MolFromSmiles(smi)atoms = mol.GetAtoms()print(type(atoms))print(type(atoms[0]))- atoms的类型为:<class ‘rdkit.Chem.rdchem._ROAtomSeq’> 可以看成是atom的列表。
- atom的类型:<class ‘rdkit.Chem.rdchem.Atom’>
获取分子中的键
GetBonds()mol对象中有获取所有键的方法
GetBonds(); - mol.GetBonds()示例:
from rdkit import Chemsmi='CC(C)OC(=O)C(C)NP(=O)(OCC1C(C(C(O1)N2C=CC(=O)NC2=O)(C)F)O)OC3=CC=CC=C3'mol = Chem.MolFromSmiles(smi)bonds = mol.GetBonds()print(type(bonds))print(type(bonds[0]))- bonds的类型为<class ‘rdkit.Chem.rdchem._ROBondSeq’>,可以看成是bond的列表。
- bond的类型为:<class ‘rdkit.Chem.rdchem.Bond’>。
根据原子编号获取键
GetAtomWithIdx()获取编号为0的原子
from rdkit import Chemsmi='CC(C)OC(=O)C(C)NP(=O)(OCC1C(C(C(O1)N2C=CC(=O)NC2=O)(C)F)O)OC3=CC=CC=C3'mol = Chem.MolFromSmiles(smi)atom0 = mol.GetAtomWithIdx(0)print(type(atom0))获取键的信息
得到键对象bond, 内置了大量的方法:
GetBondType():获取键的类型GetBeginAtom(): 组成键的第一个原子GetBeginAtomIdx(): 键的第一个组成原子编号GetEndAtomIdx():键的第二个组成原子编号GetEndAtom(): 组成键的第二个原子IsInRing(): 判断是否在环上IsInRingSize(n):判断是否在n-元环上
更多方法参见
rdkit.Chem.rdchem.Bond <https://www.rdkit.org/docs/source/rdkit.Chem.rdchem.html#rdkit.Chem.rdchem.Bond>_。注解
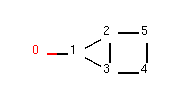
rdkit采用的是最小成环原则: 比如两个环并在一起,从数学上看可以看成是2个小环,一个大环。在rdkit中计算环的时候只考虑小环。
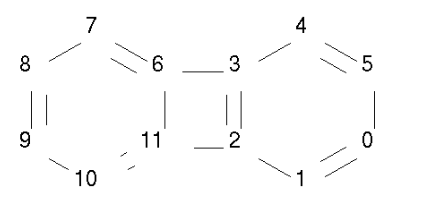
如下图所示,我们构建了一个4元环并3元环的分子;
图 分子(’OC1C2C1CC2)
我们可以看到该分子是由3元环并4元环组成的,其中原子2和原子3位于3元环和4元环的公共边上。
from rdkit import Chemm = Chem.MolFromSmiles('OC1C2C1CC2')atom2 = m.GetAtomWithIdx(2)print("atom2 in ring:",atom2.IsInRing())print("atom2 in 3-ring:",atom2.IsInRingSize(3))print("atom2 in 4-ring:",atom2.IsInRingSize(4))print("atom2 in 5-ring:",atom2.IsInRingSize(5))输出:
xxxxxxxxxxatom2 in ring: Trueatom2 in 3-ring: Trueatom2 in 4-ring: Trueatom2 in 5-ring: False获取分子中所有的环
GetSymmSSSR(m)获取分子中所有的环,以及每个环对应的原子组成。 如下图所示,我们构建了一个4元环并3元环的分子;
分子(
OC1C2C1CC2)from rdkit import Chemm = Chem.MolFromSmiles('OC1C2C1CC2')ssr = Chem.GetSymmSSSR(m)num_ring = len(ssr)print("num of ring",num_ring)for ring in ssr:print("ring consisted of atoms id:",list(ring))输出:
xxxxxxxxxxnum of ring 2ring consisted of atoms id: [1, 2, 3]ring consisted of atoms id: [4, 5, 2, 3]通过计算,我们发现示例分子一共有2个环,第一个环由3个原子(原子1、2、3)组成, 第二个环由4个原子组成(原子4,5,2,3)
获取分子中环的个数
Chem.GetSSSR(m)from rdkit import Chemm = Chem.MolFromSmiles('OC1C2C1CC2')num_ring = Chem.GetSSSR(m)print("num rings:",num_ring)输出:
xxxxxxxxxxnum rings: 2获取分子中环的信息
m.GetRingInfo()from rdkit import Chemm = Chem.MolFromSmiles('OC1C2C1CC2')ri = m.GetRingInfo()print(type(ri))ri的类型为 rdkit.Chem.rdchem.RingInfo。 RingInfo 对象中内置了多种方法:NumAtomRings(n): 原子n是否在环上;IsAtomInRingOfSize(1,3): 原子1是否在3元环上。
更多方法参见 rdkit.Chem.rdchem.RingInfo
3. 修改分子
增删H原子:
1.增加氢原子:
Chem.AddHs(); 2. 删除氢原子:Chem.RemoveHs(m2)RDKit 中的分子默认采用隐式H原子形式。 RDKit 中提供了Chem.AddHs()方法,添加H原子。
from rdkit import Chemm = Chem.MolFromSmiles('OC1C2C1CC2')m2 = Chem.AddHs(m)print("m Smiles:",Chem.MolToSmiles(m))print("m2 Smiles:",Chem.MolToSmiles(m2))print("num ATOMs in m:",m2.GetNumAtoms())print("num ATOMs in m2:",m.GetNumAtoms())输出:
xxxxxxxxxxm Smiles: OC1C2CCC12m2 Smiles: [H]OC1([H])C2([H])C([H])([H])C([H])([H])C12[H]num ATOMs in m: 14num ATOMs in m2: 63D构象优化的时候,需要采用显式H原子
芳香共轭键和库里单双键
Chem.Kekulize(m)RDKit 默认把芳香体系的键的类型识别为芳香键。
以苯为例:
m = Chem.MolFromSmiles('c1ccccc1')for bond in m.GetBonds():print(bond.GetBondType())输出:
xxxxxxxxxxAROMATICAROMATICAROMATICAROMATICAROMATICAROMATIC将芳香键的类型修改为单双键的类型
Chem.Kekulize(m)m = Chem.MolFromSmiles('c1ccccc1')Chem.Kekulize(m)for bond in m.GetBonds():print(bond.GetBondType())print("bond 1 is aromatic",m.GetBondWithIdx(1).GetIsAromatic())print("atom 1 is aromatic",m.GetAtomWithIdx(1).GetIsAromatic())输出:
xxxxxxxxxxDOUBLESINGLEDOUBLESINGLEDOUBLESINGLEbond 1 is aromatic Trueatom 1 is aromatic True尽管键的类型更改为单双键的形式,但是原子和键的本质依旧是芳香性的。
强制修改键的类型形式和本质:
Chem.Kekulize(m1, clearAromaticFlags=True)不建议使用。将键的类型恢复到芳香键的形式:
Chem.SanitizeMol(m)
4. 处理2D分子
Smiles 可以看成分子的1D形式,分子的平面结构可以看成分子的2D形式
产生2D坐标
AllChem.Compute2DCoords(m)AllChem.Compute2DCoords(m)产生固定的独一无二的取向。注解
该算法能够减少分子中原子在平面内的碰撞,使得绘制的分子更加清晰。
但对于复杂分子如青蒿素依旧不能解决平面碰撞问题。
可用于作为绘制分子的模板。
如果有多个分子共享一个骨架,我们希望绘图的时候能够保证在这些分子中的骨架方向是一致的。 首先把骨架提取成绘图模板,然后在绘图上添加基团。
举例:
含吡咯并嘧啶子结构的化合物
我们可以看到这4个结构都含有吡咯并嘧啶(c1nccc2n1ccc2)的子结构, 但是在图片上他们的子结构取向不一致,不便于比较他们的结构。 我们可以采用模板绘制法,固定公共子结构的取向。
代码:
x
from rdkit.Chem import Drawfrom rdkit import Chemsmis=['COC1=C(C=CC(=C1)NS(=O)(=O)C)C2=CN=CN3C2=CC=C3',# 'CCN(CC1=C(C=CC(=C1)C(F)(F)F)C2=CC(=C3N2C=NC=C3)CC(=O)O)C(=O)C4CC4','C1=CC2=C(C(=C1)C3=CN=CN4C3=CC=C4)ON=C2C5=CC=C(C=C5)F','COC(=O)C1=CC2=CC=CN2C=N1','C1=C2C=C(N=CN2C(=C1)Cl)C(=O)O',]template = Chem.MolFromSmiles('c1nccc2n1ccc2')AllChem.Compute2DCoords(template)mols=[]for smi in smis:mol = Chem.MolFromSmiles(smi)AllChem.GenerateDepictionMatching2DStructure(mol,template)mols.append(mol)img=Draw.MolsToGridImage(mols,molsPerRow=4,subImgSize=(200,200),legends=['' for x in mols])img输出:
这样我们就可以很清楚的看出这4个分子取代基团和位置的差异。
基于3D构象产生2D取向
AllChem.GenerateDepictionMatching3DStructure()rdkit 支持2D取向和3D取向对应。 这里以PDB CODE: 1T3R为例。我们获得其中的ligand并保存为mol文件。 经过测试发现产生的2D结构会有碰撞,该方法尚不成熟。
5. 处理3D分子
RDKit中内置了2种不同的方法产生分子构象。
方法一: 距离几何法[1]
基于链接表和规则计算分子中原子的距离矩阵;
使用三角平滑算法对距离矩阵进行优化;
产生一个随机距离矩阵;
为每个原子产生一个坐标;
使用粗糙的立场对坐标进行优化
(通过该方法产生的3D构象通常很丑陋。需要进一步通过力场进行美化。RDKit美化分子构象使用的是UFF力场)
方法二:基于CSD的扭转角偏好方法【18】 通过该方法产生的构象不需要进一步通过力场进行优化。 从2018.09版本开始,RDKit产生3D构象的默认方法是方法二ETKDG。
产生3D构象
AllChem.EmbedMolecule(m)注解
产生3D构象前,需要为分子添加H原子。
m2=Chem.AddHs(mol)加氢原子AllChem.EmbedMolecule(mol)2D->3D化m3 = Chem.RemoveHs(m2)删除氢原子
产生多个3D构象
AllChem.EmbedMultipleConfs(m2, numConfs=10):为m2分子产生10个构象,保存在m2分子中。- 访问指定构象:
m2.GetConformer(1) - 获取构象:
m2.GetConformers()
产生多个构象比较耗时,支持多线程加速。
cids = AllChem.EmbedMultipleConfs(m2,numConfs=10, numThreads=4)产生10个构象; .. code-block:: py
cids = AllChem.EmbedMultipleConfs(m2, numConfs=10) m2.GetConformers() m2.GetConformer(1)
计算构象的RMSD
- 计算其他构象和第一个构象的RMSD值
rmslist = []AllChem.AlignMolConformers(m2, RMSlist=rmslist)- 计算指定两个构象的RMSD值
rms = AllChem.GetConformerRMS(m2, 1, 9, prealigned=True)MMFF力场对构象进行优化
AllChem.MMFFOptimizeMoleculeConfsxxxxxxxxxxres = AllChem.MMFFOptimizeMoleculeConfs(m2)构象优化比较耗时,支持多线程加速。
多线程:
res = AllChem.MMFFOptimizeMoleculeConfs(m2, numThreads=0)
6. 保存分子对象
RdDkit 内置了2种保存分子对象的方法。
分子对象可以使用python的pickling机制进行保存。
pkl = pickle.dumps(m)RDKit的pickle文件非常紧凑,从pickle文件中加载分子比Mol文件和SMILES字符串快很多。 对于经常使用的分子保存为pickle 是一个好主意。
分子对象也可以保存为普通的二进制文件。binStr = m.ToBinary()
二进制文件的大小和分子大小有关系,而pickle文件和分子大小关系不明显。推荐大家用pickle保存文件。 pickle文件比sdf文件更小,加载速度更快。
7. 绘制分子【可视化分子】
这里主要介绍绘制分子的常规方法,更多自定义绘制,参见进阶教程。 rdkit 内置了Draw模块,用于绘图,把一些经常用到的方法直接放在Draw下面。
分子对象转化为图片
Draw.MolToFile(mol,’filename.png’)显示分子中原子的编号
示例代码
xxxxxxxxxxfrom rdkit import Chemfrom rdkit.Chem import Drawfrom rdkit.Chem.Draw import IPythonConsole #Needed to show moleculesfrom rdkit.Chem.Draw.MolDrawing import MolDrawing, DrawingOptions #Only needed if modifying defaultsopts = DrawingOptions()opts.includeAtomNumbers=Truem = Chem.MolFromSmiles('OC1C2C1CC2')opts.includeAtomNumbers=Trueopts.bondLineWidth=2.8Draw.MolToImage(m,options=opts)输出:
多个分子按照grid显示
示例代码
xxxxxxxxxxfrom rdkit.Chem import Drawfrom rdkit import Chemsmis=['COC1=C(C=CC(=C1)NS(=O)(=O)C)C2=CN=CN3C2=CC=C3','C1=CC2=C(C(=C1)C3=CN=CN4C3=CC=C4)ON=C2C5=CC=C(C=C5)F','COC(=O)C1=CC2=CC=CN2C=N1','C1=C2C=C(N=CN2C(=C1)Cl)C(=O)O',]mols=[]for smi in smis:mol = Chem.MolFromSmiles(smi)mols.append(mol)img=Draw.MolsToGridImage(mols,molsPerRow=4,subImgSize=(200,200),legends=['' for x in mols])img输出:
多个分子基于公共骨架按照grid显示
上述分子的公共结构,取向不一致,不便于分析。引入template作图法,可调整取向。
示例代码:
xxxxxxxxxxfrom rdkit.Chem import Drawfrom rdkit import Chemsmis=['COC1=C(C=CC(=C1)NS(=O)(=O)C)C2=CN=CN3C2=CC=C3',# 'CCN(CC1=C(C=CC(=C1)C(F)(F)F)C2=CC(=C3N2C=NC=C3)CC(=O)O)C(=O)C4CC4','C1=CC2=C(C(=C1)C3=CN=CN4C3=CC=C4)ON=C2C5=CC=C(C=C5)F','COC(=O)C1=CC2=CC=CN2C=N1','C1=C2C=C(N=CN2C(=C1)Cl)C(=O)O',]template = Chem.MolFromSmiles('c1nccc2n1ccc2')AllChem.Compute2DCoords(template)mols=[]for smi in smis:mol = Chem.MolFromSmiles(smi)AllChem.GenerateDepictionMatching2DStructure(mol,template)mols.append(mol)img=Draw.MolsToGridImage(mols,molsPerRow=4,subImgSize=(200,200),legends=['' for x in mols])img输出:
8. 子结构搜索:
子结构搜索可以通过SMARTS匹配符完成。
判断是否有子结构
m.HasSubstructMatch(patt)首先创建分子对象,然后定义匹配模式,最后判断是否有子结构。
匹配模式:支持Chem.MolFromSmarts() 和 Chem.MolFromSmiles() 两种形式。 Smarts的表现形式更加丰富。
示例代码:
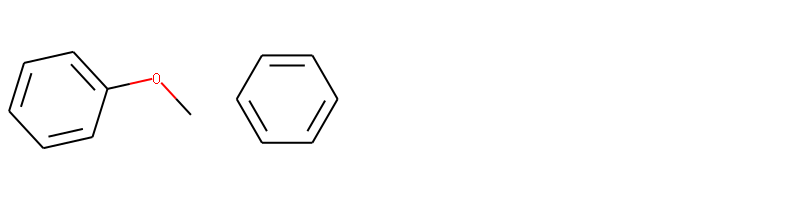
m = Chem.MolFromSmiles('c1ccccc1OC')patt = Chem.MolFromSmarts('OC')flag =m.HasSubstructMatch(patt)if flag:print("molecu m contains group -OCH3")else:print("molecu m don't contain group -OCH3")我们可以知道甲氧基苯含有甲氧基基团。
获取第一个子结构对应的原子编号
m.GetSubstructMatch()我们以甲氧基苯为例:
查找-OCH3 对应的原子编号。
代码:
xxxxxxxxxxm = Chem.MolFromSmiles('c1ccccc1OC')patt = Chem.MolFromSmarts('OC')flag =m.HasSubstructMatch(patt)if flag:atomids = m.GetSubstructMatch(patt)print("matched atom id:",atomids)else:print("molecu m don't contain group -OCH3")输出: matched atom id: (6, 7)。
从输出我们知道6号原子对应的是甲氧基原子的O原子,7号原子对应的是甲氧基原子的C原子。
注解 原子的先后顺序和SMARTS 一一对应。
获取对应所有子结构(
m.GetSubstructMatches)对相应的原子编号示例分子:
查找里面的甲氧基对应的原子编号。
m = Chem.MolFromSmiles('c1ccc(OC)cc1OC')patt = Chem.MolFromSmarts('OC')flag =m.HasSubstructMatch(patt)if flag:atomids = m.GetSubstructMatches(patt)print("matched atom id:",atomids)else:print("molecu m don't contain group -OCH3")输出: matched atom id: ((4, 5), (8, 9))
子结构搜索考虑手性
help(rdkit.Chem.rdchem.Mol.HasSubstructMatch) 默认是不考虑手性的,
useChirality=False。注解
- 不考虑手性的时候,有手性的分子可以匹配无手性的模式和错误手性的模式。
- 考虑手性的时候: 有手性信息的分子可以匹配无手性的模式; 无手性信息的分子不能匹配有手性的模式。
不考虑手性示例代码:
xxxxxxxxxxm = Chem.MolFromSmiles('CC[C@H](F)Cl')print(m.HasSubstructMatch(Chem.MolFromSmiles('C[C@H](F)Cl')))print(m.HasSubstructMatch(Chem.MolFromSmiles('C[C@@H](F)Cl')))print(m.HasSubstructMatch(Chem.MolFromSmiles('CC(F)Cl')))输出:
xxxxxxxxxxTrueTrueTrue考虑手性,示例代码:
xxxxxxxxxxm = Chem.MolFromSmiles('CC[C@H](F)Cl')a=m.HasSubstructMatch(Chem.MolFromSmiles('C[C@H](F)Cl'),useChirality=True)b=m.HasSubstructMatch(Chem.MolFromSmiles('C[C@@H](F)Cl'),useChirality=True)c=m.HasSubstructMatch(Chem.MolFromSmiles('CC(F)Cl'),useChirality=True)print(a)print(b)print(c)开启手性匹配的时候,有手性信息的分子可以匹配无手性的模式和对应手性的模式;
输出:
xxxxxxxxxxTrueFalseTrue考虑手性,示例代码:
xxxxxxxxxxm2 = Chem.MolFromSmiles('CCC(F)Cl')print(m2.HasSubstructMatch(Chem.MolFromSmiles('C[C@H](F)Cl'),useChirality=True))输出是False,说明没有手性的分子不能匹配到手性模式。
SMARTS
SMARTS 在子结构匹配、化学反应等方面发挥着重要作用。 常用的SMARTS知识:
- C c 大写小写C是不一样的,大写代表脂肪碳;小写代表芳香碳。
- 冒号后面的数字为Atom Map ID
- 羟基O的表示 []
9. 化学转换
RDKit中包含了大量修改分子的函数。 这些函数仅仅是对分子做简单的变换。 对于复杂变换参见化学反应小节。化学反应
删除子结构
AllChem.DeleteSubstructs(m,patt)创建分子对象和子结构对象,然后执行
AllChem.DeleteSubstructs(m,patt)方法, 就能得到一个删除子结构的新的分子对象。如删除甲氧基苯上面的甲氧基基团。
示例代码:
from rdkit import Chemfrom rdkit.Chem import AllChemfrom rdkit.Chem import Drawm = Chem.MolFromSmiles('c1ccccc1OC')patt = Chem.MolFromSmarts('OC')m2=AllChem.DeleteSubstructs(m,patt)mols=[m,m2]Draw.MolsToGridImage(mols,molsPerRow=4,subImgSize=(200,200),legends=['' for x in mols])输出:
我们可以删除甲氧基后得到一个新的分子苯。
取代基替换
AllChem.ReplaceSubstructs(m,patt,repl)我们利用定义一些取代基[-Cl,-Br,-OH,-NO2等],然后来替换甲氧基苯上的甲氧基。 从而能够能够产生一系列的衍生物。
代码如下:
xxxxxxxxxxfrom rdkit import Chemfrom rdkit.Chem import AllChemfrom rdkit.Chem import Drawm = Chem.MolFromSmiles('COc1c(Br)cccc1OC')patt = Chem.MolFromSmarts('OC')repsmis= ['F','Cl','Br','O']mols=[]mols.append(m)for r in repsmis:rep = Chem.MolFromSmarts(r)res = AllChem.ReplaceSubstructs(m,patt,rep)mols.extend(res)smis = [ Chem.MolToSmiles(mol) for mol in mols]mols = [Chem.MolFromSmiles(smi) for smi in smis]Draw.MolsToGridImage(mols,molsPerRow=3,subImgSize=(200,200),legends=['' for x in mols])输出:
替换操作返回的是分子对象操作列表, 如果分子只有一个地方匹配到,则返回一个分子的列表。 如果分子中有2个地方匹配到,则返回2个分子的列表。 为了标准化smiles,可以将得到的分子mol->smiles->mol,然后对其可视化。
SAR分析——core可视化
Chem.ReplaceSidechains(m1,core)Chem.ReplaceSidechains(m1,core): 我们需要定义分子对象,骨架分子; 然后执行ReplaceSidechains函数,删除侧链就能得到骨架可视化。整个过程中不需要定义侧链。
侧链+骨架=分子
我们创建了分子,如下图所示。
定义嘧啶为核心结构。通过RDKit可以对其骨架可视化。
示例代码:
xxxxxxxxxxm1 = Chem.MolFromSmiles('BrCCc1cncnc1C(=O)O')core = Chem.MolFromSmiles('c1cncnc1')tmp = Chem.ReplaceSidechains(m1,core)tmp输出:
我们可以看到嘧啶骨架上有两个地方连接侧链。
SAR分析——sidechain可视化
Chem.ReplaceCore(m1,core)Chem.ReplaceCore(m1,core): 我们需要定义分子对象,骨架分子;然后执行
Chem.ReplaceCore(m1,core)函数,删除骨架就能得到侧链。整个过程中不需要定义侧链。
侧链+骨架=分子
Chem.ReplaceCore(m1,core)我们创建了分子,如下图所示。
定义嘧啶为核心结构。通过RDKit可以对其侧链可视化。
示例代码:
xxxxxxxxxxm1 = Chem.MolFromSmiles('BrCCc1cncnc1C(=O)O')core = Chem.MolFromSmiles('c1cncnc1')tmp = Chem.ReplaceCore(m1,core)tmp输出:
注解
侧链的编号默认是从1开始,取决于算法找到侧链的先后顺序。 也可以根据侧链链接连接到骨架上的原子进行编号
tmp=Chem.ReplaceCore(m1,core,labelByIndex=True)。拆分片段
Chem.GetMolFrags(tmp,asMols=True)删除得到的侧链tmp 包含多个片段,我们可通过GetMolFrags获得里面的片段。
rs = Chem.GetMolFrags(tmp,asMols=True)对多个分子进行SAR分析
以下述结构为例,发现其公共子结构是溴苯。
计算公共子结构的方法,参见
最大公共子结构FindMCS小节。首先获取溴苯的骨架可视化。
示例代码:
xxxxxxxxxxfrom rdkit import Chemfrom rdkit.Chem import AllChemfrom rdkit.Chem import Drawm = Chem.MolFromSmiles('COc1c(Br)cccc1OC')core = Chem.MolFromSmiles('c1c(Br)cccc1')core_v = Chem.ReplaceSidechains(m,core)core_v输出:
然后对其侧链进行可视化:
多个分子的难点是取代基的编号如何统一?(统一不了的话分几个字典储存了之后加一个classification再处理呢,但仅对苯环结构可以)
获取分子骨架Murcko Decomposition
把分子中环结构提取出来,然后保留连接环结构的最少的键,如果该结构上的原子直接相连双键, 则保留双键,得到的结构称为Murcko骨架。
这里我以泽布替尼、瑞德西韦和氯胺酮为例。
我们借助Murcko算法可以自动提取里面的骨架。
示例代码:
xxxxxxxxxxfrom rdkit import Chemfrom rdkit.Chem.Scaffolds import MurckoScaffoldm1 = Chem.MolFromSmiles('C=CC(=O)N1CCC(CC1)C2CCNC3=C(C(=NN23)C4=CC=C(C=C4)OC5=CC=CC=C5)C(=O)N')m2 = Chem.MolFromSmiles('CCC(CC)COC(=O)C(C)NP(=O)(OCC1C(C(C(O1)(C#N)C2=CC=C3N2N=CN=C3N)O)O)OC4=CC=CC=C4')m3= Chem.MolFromSmiles('CNC1(CCCCC1=O)C1=CC=CC=C1Cl')core_m1 = MurckoScaffold.GetScaffoldForMol(m1)core_m2 = MurckoScaffold.GetScaffoldForMol(m2)core_m3 = MurckoScaffold.GetScaffoldForMol(m3)core_mols=[core_m1,core_m2,core_m3]Draw.MolsToGridImage(core_mols,molsPerRow=3,subImgSize=(300,300),legends=['' for x in core_mols])输出为
最大公共子结构FindMCS
RDKit内置了计算最大公共子结构的函数FindMCS, 接受 必须参数:带计算的分子列表。 可选参数:
- bondCompare 回调函数
- atomCompare 回调函数
- timeout
返回的是 rdkit.Chem.rdFMCS.MCSResult 对象。
更多细节参见 rdkit.Chem.rdFMCS
以如下3个分子为例,计算他们的最大公共子结构。
示例代码:
xxxxxxxxxxfrom rdkit.Chem import rdFMCSfrom rdkit import Chemmol1 = Chem.MolFromSmiles("O=C(NCc1cc(OC)c(O)cc1)CCCC/C=C/C(C)C")mol2 = Chem.MolFromSmiles("CC(C)CCCCCC(=O)NCC1=CC(=C(C=C1)O)OC")mol3 = Chem.MolFromSmiles("c1(C=O)cc(OC)c(O)cc1")mols = [mol1,mol2,mol3]res=rdFMCS.FindMCS(mols)common=Chem.MolFromSmarts(res.smartsString)common输出:
默认atomCompare 和 bondCompare 采用 (AtomCompare)atomCompare=rdkit.Chem.rdFMCS.AtomCompare.CompareElements 要求他们元素相同和 (BondCompare)bondCompare=rdkit.Chem.rdFMCS.BondCompare.CompareOrder 有相同的键的类型。
atomCompare也有其他的内置函数如:
- rdkit.Chem.rdFMCS.AtomCompare.CompareElements
- CompareAny = rdkit.Chem.rdFMCS.AtomCompare.CompareAny
- CompareAnyHeavyAtom = rdkit.Chem.rdFMCS.AtomCompare.CompareAnyHeavyAtom
- CompareElements = rdkit.Chem.rdFMCS.AtomCompare.CompareElements
- CompareIsotopes = rdkit.Chem.rdFMCS.AtomCompare.CompareIsotopes
bondCompare也有其他的内置函数如:
- CompareAny = rdkit.Chem.rdFMCS.BondCompare.CompareAny
- CompareOrder = rdkit.Chem.rdFMCS.BondCompare.CompareOrder
- CompareOrderExact = rdkit.Chem.rdFMCS.BondCompare.CompareOrderExac
MCS算法搜索公共子结构的通常花费几秒钟的时间,如果遇到复杂结构,通常需要几分钟甚至更长时间。 默认timeout=3600秒。如果超过默认时间,则这个res.canceled属性会被设置成True。
示例代码:
xxxxxxxxxxmols = [Chem.MolFromSmiles("Nc1ccccc1"*10), Chem.MolFromSmiles("Nc1ccccccccc1"*10)]rs=rdFMCS.FindMCS(mols, timeout=1)print(rs.canceled)print(rs.smartsString)输出:
xxxxxxxxxxTrue[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]):[#6](:[#6]:[#6])-[#7]-[#6](:[#6]:[#6]:[#6]):[#6]:[#6]到达timeout 会返回目前找到的最大子结构。
10. 指纹和相似性
RDKit 内置了多种分子指纹计算方法,如:
- 拓扑指纹 Chem.RDKFingerprint(mol)
- MACCS 指纹
- Atom Pairs
- topological torsions
- 摩根指纹(圆圈指纹)
- 摩根指纹拓展
More details about the algorithm used for the RDKit fingerprint can be found in the “RDKit Book”. RDKit中分子指纹的细节在RDkit Book中。
拓扑指纹
Chem.RDKFingerprint(x)示例代码:
ms = [Chem.MolFromSmiles('CCOC'), Chem.MolFromSmiles('CCO'), Chem.MolFromSmiles('COC')]fps = [Chem.RDKFingerprint(x) for x in ms]MACCS 指纹
MACCSkeys.GenMACCSKeys(mol)示例代码:
xxxxxxxxxxfrom rdkit.Chem import MACCSkeysfps = [MACCSkeys.GenMACCSKeys(x) for x in ms]Atom Pairs
示例代码:
xxxxxxxxxxfrom rdkit.Chem.AtomPairs import Pairsms = [Chem.MolFromSmiles('C1CCC1OCC'),Chem.MolFromSmiles('CC(C)OCC'),Chem.MolFromSmiles('CCOCC')]pairFps = [Pairs.GetAtomPairFingerprint(x) for x in ms]
topological torsions
示例代码:
from rdkit.Chem.AtomPairs import Torsionsms = [Chem.MolFromSmiles('C1CCC1OCC'),Chem.MolFromSmiles('CC(C)OCC'),Chem.MolFromSmiles('CCOCC')]tts = [Torsions.GetTopologicalTorsionFingerprintAsIntVect(x) for x in ms]摩根指纹(圆圈指纹)
AllChem.GetMorganFingerprint(mol,2)摩根指纹又称为圆圈指纹。 产生摩根指纹的时候,需要指定指纹的半径。
示例结构:
xxxxxxxxxxfrom rdkit.Chem import AllChemm1 = Chem.MolFromSmiles('Cc1ccccc1')fp1 = AllChem.GetMorganFingerprint(m1,2)基于指纹计算相似性
相似性计算方法有:
- Tanimoto, 默认的方法
- Dice,
- Cosine,
- Sokal,
- Russel,
- Kulczynski,
- McConnaughey, and
- Tversky.
比较下面3个分子的相似性。
- 分子1: CC(=O)CC(C1=CC=C(C=C1)N+=O)C1=C(O)C2=CC=CC=C2OC1=O
- 分子2: CC(=O)CC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2
- 分子3: CCC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2
方案一:基于拓扑指纹 和 Tanimoto相似性方法(默认) 指纹计算3个分子的相似性
示例代码:
from rdkit import DataStructsimport rdkitfrom rdkit import Chemfrom rdkit.Chem import Drawsmis=['CC(=O)CC(C1=CC=C(C=C1)[N+]([O-])=O)C1=C(O)C2=CC=CC=C2OC1=O','CC(=O)CC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2','CCC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2']mols =[]for smi in smis:m = Chem.MolFromSmiles(smi)mols.append(m)fps = [Chem.RDKFingerprint(x) for x in mols]sm01=DataStructs.FingerprintSimilarity(fps[0],fps[1])sm02=DataStructs.FingerprintSimilarity(fps[0],fps[2])sm12=DataStructs.FingerprintSimilarity(fps[1],fps[2])print("similarity between mol 1 and mol2: %.2f"%sm01)print("similarity between mol 1 and mol3: %.2f"%sm02)print("similarity between mol 2 and mol3: %.2f"%sm12)输出:
xxxxxxxxxxsimilarity between mol 1 and mol2: 0.93similarity between mol 1 and mol3: 0.87similarity between mol 2 and mol3: 0.93相似性表明发现分子1 和分子3 差异最大。
方案二: 基于MACCS 指纹和Dice 相似性方法计算相似性
示例代码:
from rdkit import DataStructsfrom rdkit.Chem import MACCSkeysimport rdkitfrom rdkit import Chemfrom rdkit.Chem import Drawsmis=['CC(=O)CC(C1=CC=C(C=C1)[N+]([O-])=O)C1=C(O)C2=CC=CC=C2OC1=O','CC(=O)CC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2','CCC(C1=CC=CC=C1)C1=C(O)C2=C(OC1=O)C=CC=C2']mols =[]for smi in smis:m = Chem.MolFromSmiles(smi)mols.append(m)fps = [MACCSkeys.GenMACCSKeys(x) for x in mols]sm01=DataStructs.FingerprintSimilarity(fps[0],fps[1],metric=DataStructs.DiceSimilarity)sm02=DataStructs.FingerprintSimilarity(fps[0],fps[2],metric=DataStructs.DiceSimilarity)sm12=DataStructs.FingerprintSimilarity(fps[1],fps[2],metric=DataStructs.DiceSimilarity)print("similarity between mol 1 and mol2: %.2f"%sm01)print("similarity between mol 1 and mol3: %.2f"%sm02)print("similarity between mol 2 and mol3: %.2f"%sm12)输出:
xxxxxxxxxxsimilarity between mol 1 and mol2: 0.78similarity between mol 1 and mol3: 0.70similarity between mol 2 and mol3: 0.92从相似性,我们可以看出分子2和分子3比较相似。
摩根指纹的形式:
摩根指纹和atom pairs 以及topological tosions 一样,有两种表现形式:
- counts (默认)
- bit vectors
示例代码:
from rdkit.Chem import AllChemm1 = Chem.MolFromSmiles('Cc1ccccc1')fp1_count = AllChem.GetMorganFingerprint(m1,2)fp1_bit = AllChem.GetMorganFingerprintAsBitVect(m1,2,nBits=1024)print(type(fp1_count))print(type(fp1_bit))输出:
xxxxxxxxxx<class 'rdkit.DataStructs.cDataStructs.UIntSparseIntVect'><class 'rdkit.DataStructs.cDataStructs.ExplicitBitVect'>摩根指纹->ECFP4 和 摩根指纹->FCFP4的比较
摩根指纹通过定义不同的invariants 可以输出ECFP、FCFP指纹。
ECFP、FCFP 不同点主要在于如何计算atom invariants.
-. ECFP 的atom invariants 是 连接信息 -. FCFP 的atom invariants 是 fature-based invariants
RDKit中Morgan算法支持的feature 有
ECFP4 和 FCFP4 中的4代表是摩根指纹的直径为4,半径为2。 这样默认半径为2的摩根指纹就是ECFP4指纹。 半径为2切考虑feature-based invariants 得到的指纹为FCFP4指纹。
RDKIT 计算ECFP4 和FCFP4的示例代码:
m1 = Chem.MolFromSmiles('c1ccccn1')ecfp4_mg = AllChem.GetMorganFingerprint(m1,2)fcfp4_mg = AllChem.GetMorganFingerprint(m1,2,useFeatures=True)注解
同样的两个分子分别基于ECFP4 和 FCFP4计算相似性其差别可能很大。
比较吡啶和呋喃的相似性
图 吡啶和呋喃的分子结构
示例代码:
m1 = Chem.MolFromSmiles('c1ccccn1')m2 = Chem.MolFromSmiles('c1ccco1')ecfp4_m1 = AllChem.GetMorganFingerprint(m1,2)ecfp4_m2 = AllChem.GetMorganFingerprint(m2,2)fcfp4_m1 = AllChem.GetMorganFingerprint(m1,2,useFeatures=True)fcfp4_m2 = AllChem.GetMorganFingerprint(m2,2,useFeatures=True)simi_ecfp4= DataStructs.DiceSimilarity(fp1,fp2)simi_fcfp4 =DataStructs.DiceSimilarity(ffp1,ffp2)print("the similarity of the two molecules calculated by ECFP4: %.2f"%simi_ecfp4)print("the similarity of the two molecules calculated by FCFP4: %.2f"%simi_fcfp4)输出:
xxxxxxxxxxthe similarity of the two molecules calculated by ECFP4: 0.36the similarity of the two molecules calculated by FCFP4: 0.91用户也可以自己定义atom invariants
以下面3个分子为例,讲述自定义atom invariants 计算分子指纹。
从上述结构来看,如果原子的atom invariants 是一样的,则分子1和分子2的指纹相同。 默认计算分子指纹的时候会考虑键的类型 bond order。因此 分子3 和分子1、2不同。 如果计算分子指纹的时候不考虑键的类型,则分子1、2、3的指纹相同。
示例代码:
m1 = Chem.MolFromSmiles('Cc1ccccc1')m2 = Chem.MolFromSmiles('Cc1ncncn1')m3 = Chem.MolFromSmiles('CC1CCCCC1')fp1 = AllChem.GetMorganFingerprint(m1,2,invariants=[1]*m1.GetNumAtoms())fp2 = AllChem.GetMorganFingerprint(m2,2,invariants=[1]*m2.GetNumAtoms())fp3 =AllChem.GetMorganFingerprint(m3,2,invariants=[1]*m3.GetNumAtoms())if(fp1==fp2):print("If set atom invariants are the same, the fp of moleclue 1 and 2 are the same too")if(fp1!=fp3):print("The fp of moleclue 1 and 3 are different because the bond order will be consided in the calculation of fp ")fp1 = AllChem.GetMorganFingerprint(m1,2,invariants=[1]*m1.GetNumAtoms(),useBondTypes=False)fp3 = AllChem.GetMorganFingerprint(m3,2,invariants=[1]*m3.GetNumAtoms(),useBondTypes=False)if(fp1==fp3):print("when atom invariants are the same and bond type not considered in the calculation of fp, the fp mol 1 and 3 are the same")输出:
xxxxxxxxxxIf set atom invariants are the same, the fp of moleclue 1 and 2 are the same tooThe fp of moleclue 1 and 3 are different because the bond order will be consided in the calculation of fpwhen atom invariants are the same and bond type not considered in the calculation of fp, the fp mol 1 and 3 are the same解释摩根指纹中bit的含义
ECFP4 以count 形式表示的时候是没有位数限制的。 ECFP4 以bit的形式表示的时候可以设置bit的位数,如果不设置默认是2048bit。 尽管是2048bit 但是是非常冗余的稀疏矩阵,里面大部分是0. 代码:
xxxxxxxxxxhelp(AllChem.GetMorganFingerprintAsBitVect)输出:
xxxxxxxxxxHelp on built-in function GetMorganFingerprintAsBitVect in module rdkit.Chem.rdMolDescriptors:GetMorganFingerprintAsBitVect(...)GetMorganFingerprintAsBitVect( (Mol)mol, (int)radius [, (int)nBits=2048 [, (AtomPairsParameters)invariants=[] [, (AtomPairsParameters)fromAtoms=[] [, (bool)useChirality=False [, (bool)useBondTypes=True [, (bool)useFeatures=False [, (AtomPairsParameters)bitInfo=None]]]]]]]) -> ExplicitBitVect :Returns a Morgan fingerprint for a molecule as a bit vectorC++ signature :class ExplicitBitVect * __ptr64 GetMorganFingerprintAsBitVect(class RDKit::ROMol,int [,unsigned int=2048 [,class boost::python::api::object=[] [,class boost::python::api::object=[] [,bool=False [,bool=True [,bool=False [,class boost::python::api::object=None]]]]]]])首先通过count形式计算ECFP4指纹中有效的信息
xxxxxxxxxxm = Chem.MolFromSmiles('c1cccnc1C')info={}fp = AllChem.GetMorganFingerprint(m,2,bitInfo=info)print("num of non-zero bit ",len(fp.GetNonzeroElements()))print("num of keys of info",len(info.keys()))输出:
xxxxxxxxxxnum of non-zero bit 16num of keys of info 16提示我们甲基吡啶分子(c1cccnc1C)在ECFP4指纹中最多有16个有效信息。
设置不同的nBits 计算有效信息的个数。
xxxxxxxxxxnbitss=[64,128,256,2048]for nbit in nbitss:mol = Chem.MolFromSmiles('c1cccnc1C')bi = {}fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=2, nBits=nbit,bitInfo=bi)print("num non zero bit in nBit=%d: %d"%(nbit,len(bi.keys())))输出:
xxxxxxxxxxnum non zero bit in nBit=64: 13num non zero bit in nBit=128: 15num non zero bit in nBit=256: 16num non zero bit in nBit=2048: 16对于分子甲基吡啶,当nBit设置256的时候就不会丢失信息的个数。
检查nBits=256和2048获取的指纹信息是否相同:
示例代码:
xxxxxxxxxxnbitss=[256,2048]bis=[]for nbit in nbitss:mol = Chem.MolFromSmiles('c1cccnc1C')bi = {}fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=2, nBits=nbit,bitInfo=bi)bis.append(bi)a=bis[0].values()b=bis[1].values()a=list(a)b=list(b)ab=a+bif len(set(ab))==len(a):print("fp info calculated by nBits=256 and 2048 are the same")输出:
xxxxxxxxxxfp info calculated by nBits=256 and 2048 are the same注解
不同位数算出来的相同信息对应在不同的bit上,且先后顺序顺序不一定一直。
查看这16个bit信息。
示例代码:
xxxxxxxxxxmol = Chem.MolFromSmiles('c1cccnc1C')bi = {}fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=2, nBits=256,bitInfo=bi)for v in bi.values():print(v)输出:
xxxxxxxxxx((5, 2),)((6, 0),)((1, 1), (2, 1))((3, 1),)((0, 0), (1, 0), (2, 0), (3, 0))((5, 0),)((4, 2),)((4, 0),)((2, 2),)((4, 1),)((1, 2),)((0, 2),)((6, 1),)((3, 2),)((5, 1),)这里我对第一个信息((5,2),) 和第三个信息((1, 1), (2, 1))进行解释。 里面的最小单元对应的是(atom index, radius)。 第一个信息是5号原子半径2的指纹。 第二个信息是1号原子和2原子原子半径为1的指纹。
获取指纹对应的结构
获取这3个指纹对应的结构信息
示例代码:
m = Chem.MolFromSmiles('c1cccnc1C')env = Chem.FindAtomEnvironmentOfRadiusN(m,2,5)amap={}submol25=Chem.PathToSubmol(m,env,atomMap=amap)env = Chem.FindAtomEnvironmentOfRadiusN(m,1,1)amap={}submol11=Chem.PathToSubmol(m,env,atomMap=amap)env = Chem.FindAtomEnvironmentOfRadiusN(m,1,2)amap={}submol12=Chem.PathToSubmol(m,env,atomMap=amap)mols=[submol25,submol11,submol12]Draw.MolsToGridImage(mols,molsPerRow=3,subImgSize=(300,200),legends=['' for x in mols])输出:

可视化指纹中的bit
RDKit中的拓扑指纹 Chem.RDKFingerprint 和摩根指纹 Morgan,可以通过如下函数进行可视化。
rdkit.Chem.Draw.DrawMorganBit()对摩根指纹中的bit 进行可视化。rdkit.Chem.Draw.DrawRDKitBit()对拓扑指纹中的bit 进行可视化。
可视化摩根指纹中的bit
首先查看分子摩根指纹里面的有效bit,
from rdkit.Chem import Drawfrom rdkit import Chemmol = Chem.MolFromSmiles('c1cccnc1C')bi = {}fp = AllChem.GetMorganFingerprintAsBitVect(mol,nBits=256, radius=2, bitInfo=bi)pprint(bi)输出:
xxxxxxxxxx{19: ((5, 2),),33: ((6, 0),),64: ((1, 1), (2, 1)),74: ((3, 1),),81: ((0, 0), (1, 0), (2, 0), (3, 0)),100: ((5, 0),),121: ((4, 2),),122: ((4, 0),),131: ((2, 2),),172: ((4, 1),),175: ((1, 2),),179: ((0, 2),),186: ((6, 1),),192: ((3, 2),),195: ((5, 1),),214: ((0, 1),)}我们可以看到有效bit 有19、33、64、74、…、214等。 我们对19、64、81的bit 进行可视化。
bits =[19,64,81]imgs=[]for bit in bits:mfp2_svg = Draw.DrawMorganBit(mol, bit, bi)imgs.append(mfp2_svg)def displayimgsinrow(imgs,col=4):plt.figure(figsize=(20,10))columns = colfor i, image in enumerate(imgs):ax=plt.subplot(len(imgs) / columns + 1, columns, i + 1)ax.set_axis_off()plt.imshow(image)displayimgsinrow(imgs)输出:
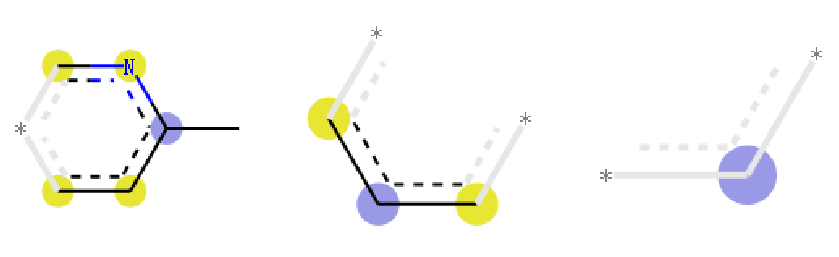
从上图中,我们可以看到对摩根指纹可视化的时候,除了可以看到片段结构, 还可以到上原子上标注了不同的颜色:
- 蓝色: 蓝色标注说明该原子是中心原子
- 黄色:说明该原子是芳香原子
- 灰色: 说明该原子时脂肪烃原子
可视化拓扑指纹中的bit
拓扑指纹也成为RDKit指纹,其调用函数Chem.RDKFingerprint(mol)
查看拓扑指纹中的有效bit。
示例代码:
xxxxxxxxxxfrom rdkit.Chem import Drawfrom rdkit import Chemmol = Chem.MolFromSmiles('c1cccnc1C')rdkbi = {}rdkfp = Chem.RDKFingerprint(mol, maxPath=2, bitInfo=rdkbi)rdkbi输出:
xxxxxxxxxx5: [[3, 4]],161: [[3, 4]],294: [[0, 1], [0, 6], [1, 2]],330: [[0, 1], [0, 6], [1, 2]],633: [[2, 3], [4, 6]],684: [[2, 3], [4, 6]],744: [[5, 6]],808: [[4, 5]],842: [[0], [1], [2], [6]],930: [[5]],1026: [[0], [1], [2], [6]],1027: [[3], [4]],1060: [[3], [4], [5]],1649: [[4, 5]],1909: [[5, 6]]}对前3个bit 进行可视化, 示例代码:
xxxxxxxxxxbits =[5,161,294]imgs=[]for bit in bits:mfp2_svg = Draw.DrawRDKitBit(mol, bit, rdkbi)imgs.append(mfp2_svg)def displayimgsinrow(imgs,col=4):plt.figure(figsize=(20,10))columns = colfor i, image in enumerate(imgs):ax=plt.subplot(len(imgs) / columns + 1, columns, i + 1)ax.set_axis_off()plt.imshow(image)displayimgsinrow(imgs)输出:

基于分子指纹挑选差异较大的分子
药物虚拟筛选中关键步骤挑选分子,比如筛选获得前1000个分子, 由于成本、时间等因素你想挑选100个分子进行活性测试, 如果你直接挑选前100个分子进行测试,命中率可能会降低。 一般流程是对1000个分子进行聚类,然后每一类里面挑选一个分子(或者中心分子), 这样可以提高分子骨架的多样性,从而提供虚拟筛选的成功率。
Schrodiger 中有这样的流程。通过RDKit 也能实现。
示例代码:
xxxxxxxxxxfrom rdkit import Chemfrom rdkit.Chem.rdMolDescriptors import GetMorganFingerprintfrom rdkit import DataStructsfrom rdkit.SimDivFilters.rdSimDivPickers import MaxMinPickerms = [x for x in Chem.SDMolSupplier('data/actives_5ht3.sdf')]while ms.count(None): ms.remove(None)fps = [GetMorganFingerprint(x,3) for x in ms]def distij(i,j,fps=fps):return 1-DataStructs.DiceSimilarity(fps[i],fps[j])picker = MaxMinPicker()pickIndices = picker.LazyPick(distij,nfps,10,seed=23)picks = [ms[x] for x in pickIndices]相似性地图
相似性地图可用于可视化原子对两个分子的相似性的贡献, 该方法位于 rdkit.Chem.Draw.SimilarityMaps 模块中。
该方法支持三种类型的指纹:
- atom pairs 类型表现形式 normal(default)、hashed 和 bit vector(bv)
- topological torsions 类型表现形式normal(default)、hashed 和 bit vector(bv)
- Morgan fingerprints 类型表现形式 bit vector(bv,default) 和 count vector(count)
示例代码:
from rdkit.Chem import Drawfrom rdkit.Chem.Draw import SimilarityMapsfp = SimilarityMaps.GetAPFingerprint(mol, fpType='normal')fp = SimilarityMaps.GetTTFingerprint(mol, fpType='normal')fp = SimilarityMaps.GetMorganFingerprint(mol, fpType='bv')计算目标相似性地图,最少需要3个参数:
- 参考分子
- 目标分子
- 指纹函数
- 相似性函数(默认是 Dice similarity)
示例代码:
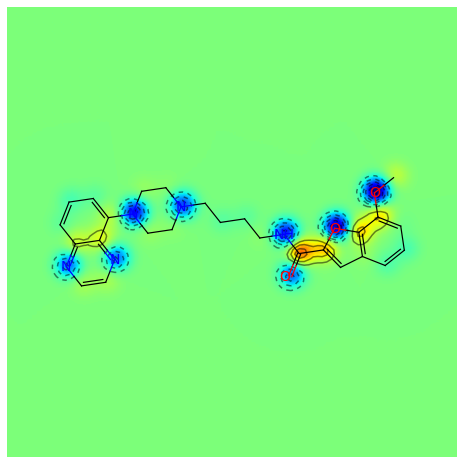
from rdkit import Chemfrom rdkit.Chem.Draw import SimilarityMapstargetmol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21')refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')target_mol_simi_fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, targetmol, SimilarityMaps.GetMorganFingerprint)输出:
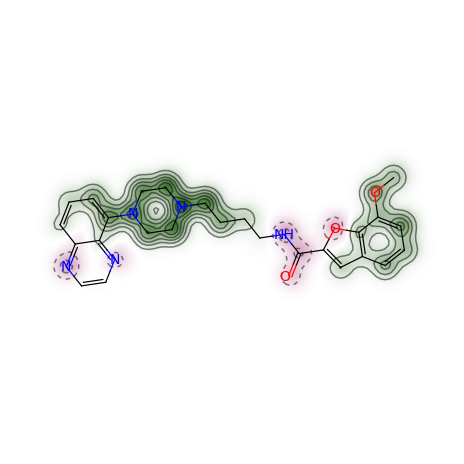
原子颜色越绿,对相似性的贡献越大。
计算相似性地图的时候,定义指纹计算方法中的参数 和相似性计算方法
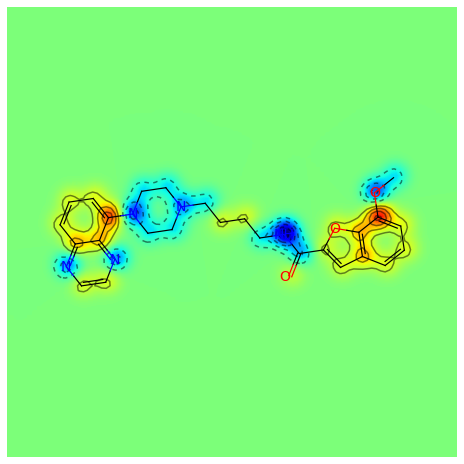
示例代码:
from rdkit import DataStructsfrom rdkit import Chemfrom rdkit.Chem.Draw import SimilarityMapstargetmol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21')refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')fig, maxweight = SimilarityMaps.GetSimilarityMapForFingerprint(refmol, mol, lambda m,idx: SimilarityMaps.GetMorganFingerprint(m, atomId=idx, radius=1, fpType='count'), metric=DataStructs.TanimotoSimilarity)输出:
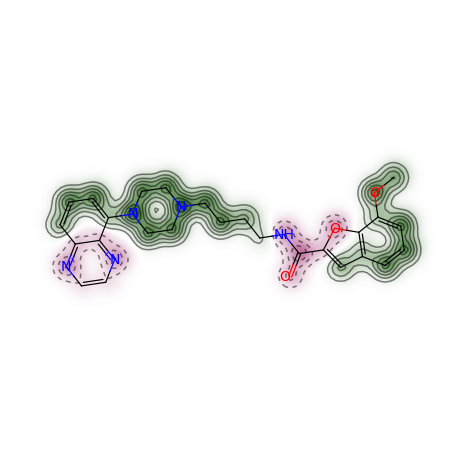
计算相似性的流程,首先计算原子的贡献,然后基于贡献值进行绘图。 因此也可以采用下述方式进行计算绘图。
示例代码:
from rdkit import DataStructsfrom rdkit import Chemfrom rdkit.Chem.Draw import SimilarityMapstargetmol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21')refmol = Chem.MolFromSmiles('CCCN(CCCCN1CCN(c2ccccc2OC)CC1)Cc1ccc2ccccc2c1')weights = SimilarityMaps.GetAtomicWeightsForFingerprint(refmol, mol, SimilarityMaps.GetMorganFingerprint)fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, weights)
11. 分子性质(描述符)
性质描述符计算
分子性质也被称为描述符。 RDKit中内置了大量的分子描述符的计算方法, 这些方法主要位于
rdkit.Chem.Descriptors <https://www.rdkit.org/docs/source/rdkit.Chem.Descriptors.html#module-rdkit.Chem.Descriptors>_ 也有些常用的性质在AllChem模块下面。RDKit 可以计算多种分子性质,如:
Descriptor/Descriptor Family Notes Gasteiger/Marsili Partial Charges Tetrahedron 36:3219-28 (1980) BalabanJ Chem. Phys. Lett. 89:399-404 (1982) BertzCT J. Am. Chem. Soc. 103:3599-601 (1981) Ipc J. Chem. Phys. 67:4517-33 (1977) HallKierAlpha Rev. Comput. Chem. 2:367-422 (1991) Kappa1 - Kappa3 Rev. Comput. Chem. 2:367-422 (1991) Chi0, Chi1 Rev. Comput. Chem. 2:367-422 (1991) Chi0n - Chi4n Rev. Comput. Chem. 2:367-422 (1991) Chi0v - Chi4v Rev. Comput. Chem. 2:367-422 (1991) MolLogP Wildman and Crippen JCICS 39:868-73 (1999) MolMR Wildman and Crippen JCICS 39:868-73 (1999) MolWt ExactMolWt HeavyAtomCount HeavyAtomMolWt NHOHCount NOCount NumHAcceptors NumHDonors NumHeteroatoms NumRotatableBonds NumValenceElectrons NumAmideBonds Num{Aromatic,Saturated,Aliphatic}Rings Num{Aromatic,Saturated,Aliphatic}{Hetero,Carbo}cycles RingCount FractionCSP3 NumSpiroAtoms Number of spiro atoms (atoms shared between rings that share exactly one atom) NumBridgeheadAtoms Number of bridgehead atoms (atoms shared between rings that share at least two bonds) TPSA J. Med. Chem. 43:3714-7, (2000) See the section in the RDKit book describing differences to the original publication. LabuteASA J. Mol. Graph. Mod. 18:464-77 (2000) PEOE_VSA1 - PEOE_VSA14 MOE-type descriptors using partial charges and surface area contributions http://www.chemcomp.com/journal/vsadesc.htm SMR_VSA1 - SMR_VSA10 MOE-type descriptors using MR contributions and surface area contributions http://www.chemcomp.com/journal/vsadesc.htm SlogP_VSA1 - SlogP_VSA12 MOE-type descriptors using LogP contributions and surface area contributions http://www.chemcomp.com/journal/vsadesc.htm EState_VSA1 - EState_VSA11 MOE-type descriptors using EState indices and surface area contributions (developed at RD, not described in the CCG paper) VSA_EState1 - VSA_EState10 MOE-type descriptors using EState indices and surface area contributions (developed at RD, not described in the CCG paper) MQNs Nguyen et al. ChemMedChem 4:1803-5 (2009) Topliss fragments implemented using a set of SMARTS definitions in $(RDBASE)/Data/FragmentDescriptors.csv Autocorr2D New in 2017.09 release. Todeschini and Consoni “Descriptors from Molecular Geometry” Handbook of Chemoinformatics https://doi.org/10.1002/9783527618279.ch37 计算分子的The topological polar surface area (TPSA) descriptor 、logP、电荷等性质,示例代码:
xxxxxxxxxxfrom rdkit.Chem import Descriptorsm = Chem.MolFromSmiles('c1ccccc1C(=O)O')tpsa_m=Descriptors.TPSA(m)logp_m=Descriptors.MolLogP(m)AllChem.ComputeGasteigerCharges(m)charge_atm0=float(m.GetAtomWithIdx(0).GetProp('_GasteigerCharge'))print("the TPSA of m is",tpsa_m)print("the logP of m is",logp_m)print("the gasteigerCharge of the first atom",charge_atm0)输出:
xxxxxxxxxxthe TPSA of m is 37.3the logP of m is 1.3848the gasteigerCharge of the first atom -0.04769375004654255原子对性质的贡献可视化
相似性地图也可用用于性质的可视化,只要性质可以分解到原子上就可以进行可视化。
Gasteiger partial 电荷的可视化距离,自定义颜色主题jet。
示例代码:
xxxxxxxxxxfrom rdkit.Chem.Draw import SimilarityMapsmol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21')AllChem.ComputeGasteigerCharges(mol)contribs = [float(mol.GetAtomWithIdx(i).GetProp('_GasteigerCharge')) for i in range(mol.GetNumAtoms())]fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, contribs, colorMap='jet', contourLines=10)输出:
RDKit中内置Crippen方法对原子logP的计算。
示例代码
xxxxxxxxxxfrom rdkit.Chem import rdMolDescriptorsmol = Chem.MolFromSmiles('COc1cccc2cc(C(=O)NCCCCN3CCN(c4cccc5nccnc54)CC3)oc21')contribs = rdMolDescriptors._CalcCrippenContribs(mol)fig = SimilarityMaps.GetSimilarityMapFromWeights(mol,[x for x,y in contribs], colorMap='jet', contourLines=10)输出:
12. 化学反应
RDKit 提供化学反应引擎,其中化学反应模板是基于smarts 构建的。 反应物+反应引擎就可以生成产物。
化学反应示例
示例反应:
反应模板,如下图所示:
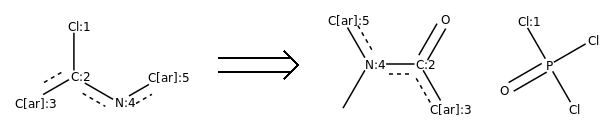
反应模板([Cl;H0;D1;+0:1]-c;H0;D3;+0:2:[n;H0;D2;+0:4]:[c:5])>>(C-n;H0;D3;+0:4:c;H0;D3;+0:2:[c:3]).(Cl-P(-Cl)(=O)-[Cl;H0;D1;+0:1])
从反应模板中,我们看到主要的变化是Cl变成羰基氧,N上多了一个甲基。
注解
这是一个逆反应模板。
反应物,如下图所示:
‘CC(C)(Nc1nc(Cl)c(-c2ccc(F)cc2)c(-c2ccncc2)n1)c1ccccc1’
- 创建具体反应规则的引擎对象 rxn = AllChem.ReactionFromSmarts(tem)
- 输入反应物,借助引擎产生反应 rxn.RunReactants([productmol])
反应示例:
xxxxxxxxxxfrom rdkit.Chem import AllChemdef getrxns(rxn,productsmi):productmol = Chem.MolFromSmiles(productsmi)reactions = rxn.RunReactants([productmol])rxns = []for reaction in reactions:smis=[]for compound in reaction:smi = Chem.MolToSmiles(compound)smis.append(smi)rxnstr='.'.join(smis)+'>>'+productsminewr=canon_reaction(rxnstr)rxns.append(newr)return rxnstem='([Cl;H0;D1;+0:1]-[c;H0;D3;+0:2](:[c:3]):[n;H0;D2;+0:4]:[c:5])>>(C-[n;H0;D3;+0:4](:[c:5]):[c;H0;D3;+0:2](=O):[c:3]).(Cl-P(-Cl)(=O)-[Cl;H0;D1;+0:1])'rxn = AllChem.ReactionFromSmarts(tem)productsmi='CC(C)(Nc1nc(Cl)c(-c2ccc(F)cc2)c(-c2ccncc2)n1)c1ccccc1'reactions =getrxns(rxn,products_smi[0])for reaction in reactions:img=ReactionStringToImage(reaction)display(img)输出:
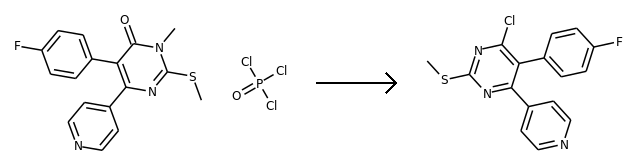
化学反应模板
化学反应模板主要可通过两种方法获取:1. 自动抽取;2. 化学家编码。
自动提取 软件:RDKit中没有提供自动提取反应模板的方法。 ASKCOS 开源了自动提取模板的方法。 原理就是化学环境发生变化的原子定义为反应中心,然后基于反应中心拓展一定的半径或者基团。
对反应模板进行高亮显示:
化学家编码 化学家通过对反应进行归纳,整理出的反应规则,典型代表Chemtic 软件(未检索到有此软件)。
化学反应注意事项
利用化学反应引擎产生的产物,不一定符合化学规则,因此需要进行对产物进行检查。
示例:
from rdkit.Chem import AllChemtem='([Cl;H0;D1;+0:1]-[c;H0;D3;+0:2](:[c:3]):[n;H0;D2;+0:4]:[c:5])>>(C-[n;H0;D3;+0:4](:[c:5]):[c;H0;D3;+0:2](=O):[c:3]).(Cl-P(-Cl)(=O)-[Cl;H0;D1;+0:1])'rxn = AllChem.ReactionFromSmarts(tem)def getrxns_reactants(rxn,productsmi):productmol = Chem.MolFromSmiles(productsmi)reactions = rxn.RunReactants([productmol])rxns = []for reaction in reactions:smis=[]for compound in reaction:# display(compound)smi = Chem.MolToSmiles(compound)smis.append(smi)newr='.'.join(smis)rxns.append(newr)return rxnsprosmi="COC(=O)c1cccc(-c2nc(Cl)cc3c(OC)nc(C(C)C)n23)c1"# prosmi='CC(C)(Nc1nc(Cl)c(-c2ccc(F)cc2)c(-c2ccncc2)n1)c1ccccc1'rs=getrxns_reactants(rxn,prosmi)smi=rs[0]m = Chem.MolFromSmiles(smi,sanitize=False)if m is None:print('invalid SMILES')else:try:Chem.SanitizeMol(m)print("smiles is ok")except:print('invalid chemistry')输出: invalid SMILES
化学反应高级功能:
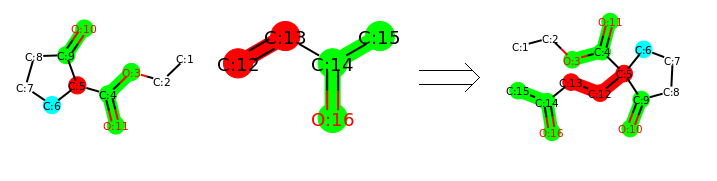
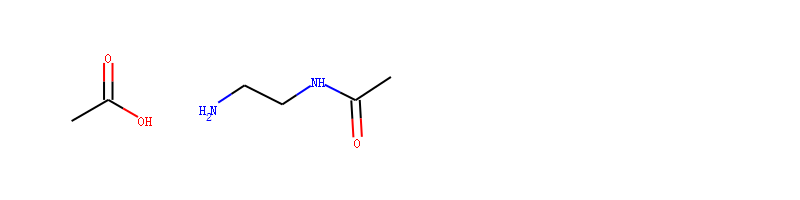
保护原子
我们以酰胺反应为例,一个分子中f含有-COOH;另一个分子中含有2个-NH。 我们定义酰胺反应模板,然后让其反应。
反应物如下图所示:
反应模板如下图所示:
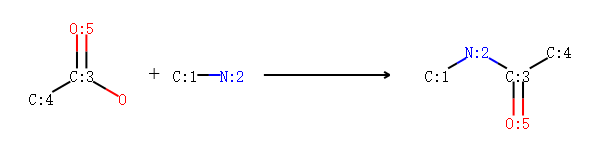
示例代码:
xxxxxxxxxxrxn = AllChem.ReactionFromSmarts('data/AmideBond.rxn')acid = Chem.MolFromSmiles('CC(=O)O')base = Chem.MolFromSmiles('CC(=O)NCCN')ps = rxn.RunReactants((acid,base))print("the number of reactions: ",len(ps))for reaction in ps:reactants_smi = Chem.MolToSmiles(reaction)print("this reaction is",reactants_smi)### 查看产物>>> Chem.MolToSmiles(ps[0][0])'CC(=O)N(CCN)C(C)=O'>>> Chem.MolToSmiles(ps[1][0])'CC(=O)NCCNC(C)=O'###输出:
xxxxxxxxxxthe number of reactions: 2如果不定义保护原子的话,则2个N都有可能参与反应。
通过指定保护原子,不允许这些原子作为反应中心发生反应。 设置属性 ‘_protected’为1就可以保护该原子。
示例代码:
xxxxxxxxxxamidep = Chem.MolFromSmarts('[N;$(NC=[O,S])]')for match in base.GetSubstructMatches(amidep):base.GetAtomWithIdx(match[0]).SetProp('_protected','1')ps = rxn.RunReactants((acid,base))print("the number of reactions: ",len(ps))输出:
xxxxxxxxxxthe number of reactions: 1把分子切成片段—— Recap 方法
Recap 方法通过对实验室中常用的化学反应来模拟化学转化将分子分子分解成一些合理的片段。 Recap 方法返回的是数据类似于node tree的结构的数据。
注解
分子片段的Smiles前面通常会加一个* 如 ‘*c1ccccc1’
>>> from rdkit import Chem>>> from rdkit.Chem import Recap>>> m = Chem.MolFromSmiles('c1ccccc1OCCOC(=O)CC')>>> hierarch = Recap.RecapDecompose(m)>>> type(hierarch)<class 'rdkit.Chem.Recap.RecapHierarchyNode'>xxxxxxxxxx>>> hierarch.smiles'CCC(=O)OCCOc1ccccc1'xxxxxxxxxx>>> ks=hierarch.children.keys()>>> sorted(ks)['*C(=O)CC', '*CCOC(=O)CC', '*CCOc1ccccc1', '*OCCOc1ccccc1', '*c1ccccc1']xxxxxxxxxx>>> ks=hierarch.GetLeaves().keys()>>> ks=sorted(ks)>>> ks['*C(=O)CC', '*CCO*', '*CCOc1ccccc1', '*c1ccccc1']xxxxxxxxxx>>> leaf = hierarch.GetLeaves()[ks[0]]>>> Chem.MolToSmiles(leaf.mol)'*C(=O)CC'把分子切成片段——BRICS 方法
RDKit 还提供了另一种把分子切成片段的方法——BRICS方法。 BRICS方法主要是根据可合成的的键对分子进行切断,因此其返回的数据结构是来自于该分子的不同分子片段, 虚拟原子(*)是告诉我们是如何切断的。
示例分子
我们对该分子进行BRICS分解,
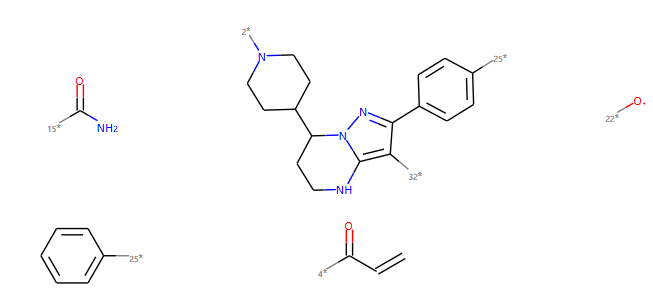
分子泽布替尼(C=CC(=O)N1CCC(CC1)C2CCNC3=C(C(=NN23)C4=CC=C(C=C4)OC5=CC=CC=C5)C(=O)N)
代码如下:
from rdkit.Chem import BRICSsmi='C=CC(=O)N1CCC(CC1)C2CCNC3=C(C(=NN23)C4=CC=C(C=C4)OC5=CC=CC=C5)C(=O)N'm=Chem.MolFromSmiles(smi)frags =(BRICS.BRICSDecompose(m))mols=[]for fsmi in frags:mols.append(Chem.MolFromSmiles(fsmi))Draw.MolsToGridImage(mols,molsPerRow=3,subImgSize=(200,200),legends=['' for x in mols])输出:
组合分子片段——BRICS方法
以上述片段进行BRICS组合产生分子,
示例代码:
xxxxxxxxxxnewms = BRICS.BRICSBuild(mols)newms=list(newms)len(newms)大概可以产生76个分子(含少量化学结构不合理的结构)
对其中前6个新组合的分子进行可视化,如图
自定义片段产生的方法
除了上面提到的自动片段分解方法,RDKit提供了更灵活的函数可根据用户定义的键进行切断产生片段。
比如对所有环上的原子和非环上的原子组成的键进行进行切断。
我们对要断开的键进行了标红处理(子结构搜索后绘制的分子图片会自动把匹配的子结构标红),如上图所示。
示例代码:
smi='C=CC(=O)N1CCC(CC1)C2CCNC3=C(C(=NN23)C4=CC=C(C=C4)OC5=CC=CC=C5)C(=O)N'm=Chem.MolFromSmiles(smi)submols = m.GetSubstructMatches(Chem.MolFromSmarts('[!R][R]'))bonds_id = [m.GetBondBetweenAtoms(x,y).GetIdx() for x,y in submols]frags = Chem.FragmentOnBonds(m,bonds_id)type(frags)smis= Chem.MolToSmiles(frags)smis=smis.split('.')mols=[]for smi in smis:mols.append(Chem.MolFromSmiles(smi))Draw.MolsToGridImage(mols,molsPerRow=3,subImgSize=(400,400),legends=['' for x in mols],useSVG=True)输出:
13. 化学性质和药效团
化学性质:
为了要得到分子的化学性质,你只要建立一个化学性质对象。
示例代码:
from rdkit import Chemfrom rdkit.Chem import ChemicalFeaturesfrom rdkit import RDConfigimport osfdefName = os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')factory = ChemicalFeatures.BuildFeatureFactory(fdefName)smi='C=CC(=O)N1CCC(CC1)C2CCNC3=C(C(=NN23)C4=CC=C(C=C4)OC5=CC=CC=C5)C(=O)N'm=Chem.MolFromSmiles(smi)feats = factory.GetFeaturesForMol(m)len(feats)for f in feats:print(f.GetFamily(),f.GetType(),f.GetAtomIds())输出:
xxxxxxxxxxDonor SingleAtomDonor (4,)Donor SingleAtomDonor (13,)Donor SingleAtomDonor (34,)Acceptor SingleAtomAcceptor (3,)Acceptor SingleAtomAcceptor (17,)Acceptor SingleAtomAcceptor (25,)Acceptor SingleAtomAcceptor (33,)Aromatic Arom5 (14, 15, 16, 17, 18)Aromatic Arom6 (19, 20, 21, 22, 23, 24)Aromatic Arom6 (26, 27, 28, 29, 30, 31)Hydrophobe ThreeWayAttach (7,)Hydrophobe ThreeWayAttach (15,)Hydrophobe ThreeWayAttach (19,)Hydrophobe ChainTwoWayAttach (1,)LumpedHydrophobe RH6_6 (19, 20, 21, 22, 23, 24)LumpedHydrophobe RH6_6 (26, 27, 28, 29, 30, 31)2D 药效团指纹
对上面计算的性质进行组合可以用作分子的2D药效团。 药效团可以进一步转化为分子药效团指纹。
示例代码
from rdkit import Chemfrom rdkit.Chem import ChemicalFeatures# fdefName = 'MinimalFeatures.fdef'# featFactory = ChemicalFeatures.BuildFeatureFactory(fdefName)fdefName = os.path.join(RDConfig.RDDataDir,'BaseFeatures.fdef')featFactory = ChemicalFeatures.BuildFeatureFactory(fdefName)from rdkit.Chem.Pharm2D.SigFactory import SigFactorysigFactory = SigFactory(featFactory,minPointCount=2,maxPointCount=3)sigFactory.SetBins([(0,2),(2,5),(5,8)])sigFactory.Init()sigFactory.GetSigSize()from rdkit.Chem.Pharm2D import Generatemol = Chem.MolFromSmiles('OCC(=O)CCCN')fp = Generate.Gen2DFingerprint(mol,sigFactory)print("figerprint size",fp.GetNumBits())print("figerprint string top 10 bit",fp.ToBitString()[0:11])输出:
xxxxxxxxxxfigerprint size 2988figerprint string top 10 bit 01000001101查看有效bit及其含义
药效团指纹和ECFP4指纹一样也是稀疏向量,大部分位置都是0。 有效bit指的是位置不是0的bit。
示例代码:
xxxxxxxxxxprint("bit==1 have these position",list(fp.GetOnBits()))print("1 postion bit",sigFactory.GetBitDescription(1))print("7 postion bit",sigFactory.GetBitDescription(7))print("0 postion bit",sigFactory.GetBitDescription(0))print("2223 postion bit",sigFactory.GetBitDescription(2223))输出:
xxxxxxxxxxbit==1 have these position [1, 7, 8, 10, 20, 47, 49, 59, 63, 73, 171, 191, 267, 488, 503, 511, 579, 622, 698, 1687, 1798, 1874, 2223]1 postion bit Acceptor Acceptor |0 1|1 0|7 postion bit Acceptor Donor |0 1|1 0|0 postion bit Acceptor Acceptor |0 0|0 0|2223 postion bit Hydrophobe Hydrophobe PosIonizable |0 0 1|0 0 1|1 1 0|
14. 分子片段
RDKit中包含了大量把分子分解成片段的方法和处理片段的工具。
RDKit中内置了一些重要的化学官能团,
示例代码:
xxxxxxxxxxfName=os.path.join(RDConfig.RDDataDir,'FunctionalGroups.txt')from rdkit.Chem import FragmentCatalogfparams = FragmentCatalog.FragCatParams(1,6,fName)fparams.GetNumFuncGroups()mols=[]for i in range(fparams.GetNumFuncGroups()):mols.append(fparams.GetFuncGroup(i))Draw.MolsToGridImage(mols,molsPerRow=8)
输出:
然后基于这份官能团列表,我们就可以分析分子中有多少个官能团。
代码如下:
xxxxxxxxxxfcat=FragmentCatalog.FragCatalog(fparams)fcgen=FragmentCatalog.FragCatGenerator()m = Chem.MolFromSmiles('OCC=CC(=O)O')fcgen.AddFragsFromMol(m,fcat)num_entries=fcat.GetNumEntries()print("matched the function group ids is",list(fcat.GetEntryFuncGroupIds(num_entries-1)))fg1=fparams.GetFuncGroup(1)fg34=fparams.GetFuncGroup(34)print("name of group 1 ",fg1.GetProp('_Name'))print("name of group 34 ",fg34.GetProp('_Name'))mols=[fg1,fg34]Draw.MolsToGridImage(mols,molsPerRow=2)
输出:
xxxxxxxxxxmatched the function group ids is [34, 1]name of group 1 -C(=O)Oname of group 34 -O
R基团分解
RDKit中自动提取R基团的算法 rdRGroupDecomposition。使用起来非常方便。
示例代码:
xfrom rdkit import Chemfrom rdkit.Chem import rdRGroupDecomposition as rdRGDsmis=['OC(C1=CC(O)=CC=C1)=O','OC(C2=CC(F)=CC=C2)=O','OC(C3=C(Br)C=CC=C3)=O']mols=[]for smi in smis:mols.append(Chem.MolFromSmiles(smi))print("number of mols",len(mols))core = Chem.MolFromSmarts('[*:1]c1cc([*:2])ccc1')res,unmatched = rdRGD.RGroupDecompose([core],mols,asSmiles=True)res
输出:
xxxxxxxxxx[{'Core': 'c1cc([*:1])c([*:3])c([*:2])c1','R1': 'O=C(O)[*:1]','R2': 'O[*:2]','R3': '[H][*:3]'},{'Core': 'c1cc([*:1])c([*:3])c([*:2])c1','R1': 'O=C(O)[*:1]','R2': 'F[*:2]','R3': '[H][*:3]'},{'Core': 'c1cc([*:1])c([*:3])c([*:2])c1','R1': 'O=C(O)[*:1]','R2': '[H][*:2]','R3': 'Br[*:3]'}]
对结果进行可视化。
15. 编辑分子
编辑实时(in-place)发生。如果希望得到合理的化学结构,一定要记得sanitize检查和修正。
如把苯环上的第一个C原子修改成N原子。
示例代码
xxxxxxxxxxm = Chem.MolFromSmiles('c1ccccc1')print("修改前的分子:")display(m)m.GetAtomWithIdx(0).SetAtomicNum(7)Chem.SanitizeMol(m)print("修改后的分子:")display(m)
输出:
xxxxxxxxxx修改前的分子:
Chapter 4 Other Tricks
Chem 和AllChem的区别
Chem: 负责基础常用的化学功能(如:读写分子,子结构搜索,分子美化等)。 AllChem: 负责高级但不常用的化学功能。 区分它们的目的是为了加速载入的速度。 如果你觉得区分它们比较麻烦,你可以使用import as 语法对它们进行合并。
xxxxxxxxxxfrom rdkit.Chem import AllChem as Chem芳香性
芳香性是一个既简单又复杂的问题。 不管是实验化学家还是理论化学家都无法给出芳香性的准确且唯一的定义。
RDKit 除了通过模式匹配已知的芳香体系进行识别,还定义一些识别芳香体系的规则。 这些规则非常直观:
- 芳香性仅仅和环上的原子和键有关。
- 芳香性键必须是由2个芳香性原子组成的。
- 由2个芳香性原子组成键不一定就是芳香键。
我们绘制示例分子,如下
xxxxxxxxxxfrom rdkit.Chem import Drawfrom rdkit.Chem.Draw import IPythonConsole #Needed to show moleculesfrom rdkit.Chem.Draw.MolDrawing import MolDrawing, DrawingOptions #Only needed if modifying defaultsopts = DrawingOptions()opts.atomLabelFontSize=400opts.includeAtomNumbers=Trueopts.dblBondLengthFrac=0.8opts.includeAtomNumbers=Trueopts.dotsPerAngstrom=1000Draw.MolToImage(m,options=opts,size=(500, 500))结构如图
基于RDKit芳香模型判断其上的原子和键的芳香性
xxxxxxxxxxarom_atom3=m.GetAtomWithIdx(3).GetIsAromatic()arom_atom6=m.GetAtomWithIdx(6).GetIsAromatic()bond_36=m.GetBondBetweenAtoms(3,6).GetIsAromatic()print("atom 3 aromatic is",arom_atom3)print("atom 6 aromatic is",arom_atom6)print("bond 36 aromatic is",bond_36)输出:
xxxxxxxxxxatom 3 aromatic is Trueatom 6 aromatic is Truebond 36 aromatic is FalseRDKit 芳香模型
芳香性是针对环体系(含融和环体系)原子和键而言的,如果该环上的pi电子满足4N+2规则,则组成该环的原子和键具有芳香性。
PI电子的计算根据原子的类型和环境具体,如下表所示。
匹配模式 pi electrons 数目 解释 c(a)a 1 芳香碳原子,链接两个芳香原子,则该碳原子贡献一个PI电子 n(a)a 1 芳香氮原子,链接两个芳香原子,则该氮原子贡献一个PI电子 An(a)a 2 芳香氮原子,链接2个芳香原子和1个任意原子的时候,贡献2个PI电子 o(a)a 2 芳香氧原子,链接2个芳香原子,贡献2个PI电子 s(a)a 2 同上 se(a)a 2 同上 te(a)a 2 同上 O=c(a)a 0 芳香碳原子链接2个芳香原子,同时参与形成羰基,贡献0个PI电子 N=c(a)a 0 同上 *(a)a 0, 1, or 2 a: 芳香原子 A: 任何原子,包含H *: 虚拟原子
知道上面的规则后,我们对下面5个分子进行判断环的芳香性。
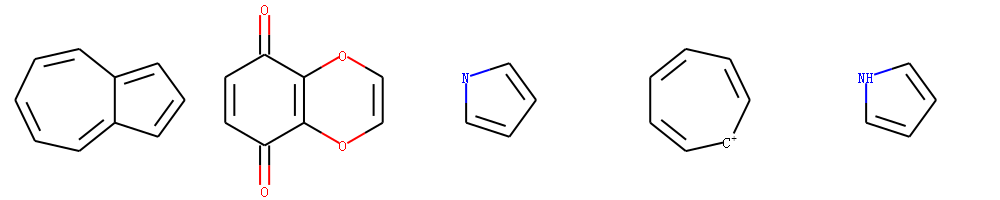
示例代码:
xxxxxxxxxxfor id ,mol in enumerate(mols):rinfo= mol.GetRingInfo()num =rinfo.NumRings()print("The %s mol has %s ring"%(id,num))rings_bond = rinfo.BondRings()bondsobj =mol.GetBonds()# print(bonds)arominfo=''for bonds in rings_bond:flag=Truefor bondid in bonds:if not bondsobj[bondid].GetIsAromatic():flag=Falsebreakarominfo+=str(flag)+" "print("arom of rings",arominfo)输出:
xxxxxxxxxxThe 0 mol has 2 ringarom of rings False FalseThe 1 mol has 2 ringarom of rings False FalseThe 2 mol has 1 ringarom of rings FalseThe 3 mol has 1 ringarom of rings FalseThe 4 mol has 1 ringarom of rings True注解
注意第一个分子虽然两个环不是芳香性的,但是整体是芳香性的。
对第一个分子进行芳香键进行匹配着色。
简单的芳香模型体系
仅仅考虑5元环或者6元环的芳香性。其电子贡献匹配模式和上面的列表一致。
MDL芳香模型体系
MDL 芳香模型体系的定义没有公开的文档对其进行说明, 唯一的介绍就是`oechem 文档 https://docs.eyesopen.com/toolkits/python/oechemtk/aromaticity.html
MDL芳香模型体系,有如下特点:
- 融合环也可能具有芳香性 ,如甘菊环分子,大的融合环有芳香性,两个小环没有芳香性。
- 五元环没有芳香性
- 只有C和N 原子具有芳香性
- 只接受原子提供一个PI电子的原子为芳香原子
- 原子在环外连接双键,则该原子没有芳香性 atoms with exocyclic double bonds are not aromatic
注解:为了方便计算,对于融合环的芳香性判断,其原子最多只允许有24个原子。
SMILES 支持和扩展:
RDKit 包含了Daylight SMILES的所有标准形式以及有用的拓展形式。
下面是部分有用的拓展。
芳香性
Te 元素(和氧同族的元素)也可能具有芳香性,当其连接2个芳香原子时候,它贡献2个PI电子。
示例分子(碲吩-2甲酸分子)
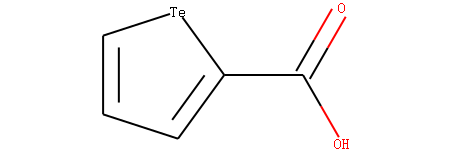
Te 原子的编号是4,我们查看其芳香性。
xxxxxxxxxxm = Chem.MolFromSmiles('OC(=O)c1[te]ccc1')arom_atom4=m.GetAtomWithIdx(4).GetIsAromatic()print("atom4 is aromatic",arom_atom4)输出:
xxxxxxxxxxatom4 is aromatic True
如何在Jupyter NoteBook 中直接显示分子对象为结构图片
xxxxxxxxxxfrom rdkit.Chem.Draw import IPythonConsole #Needed to show molecules
RDKit如何给复杂分子绘制漂亮的2D结构?
建议更新RDKit版本,同时指定开启新的2D坐标产生的算法。 默认采用的是schrodinger中的2D 算法。
xxxxxxxxxxfrom rdkit.Chem import rdDepictorrdDepictor.SetPreferCoordGen(True)Chem.MolFromSmiles('CC1CCC2C(C(=O)OC3C24C1CCC(O3)(OO4)C)C')
Chapter 5 OpenBabel Tutorial
Ref. Website: http://openbabel.org/wiki/Main_Page
Installation
Windows:
Open Babel GUI
Provides a graphical user interface for Open Babel, as well as a command-line interface.
Get the latest installer for 64-bit (recommended) or 32-bit (has "-x86" in name).
Python module (requires OpenBabelGUI above)
Provides access to the Open Babel libraries from Python
Java library (requires OpenBabelGUI above)
Provides access to the Open Babel libraries from Java.
OBDotNet assembly (requires OpenBabelGUI above)
Provides access to the Open Babel libraries from .NET languages.
The following options are only recommended for experienced developers.
Compile from source:
Download the source for the latest release
or Get latest development code
Open Babel can be compiled using any of MSVC++, Cygwin or MinGW
Linux:
Compile from source
Compile Open Babel:
or Get latest development code (today)
How to use obabel
Install a binary package
Several Linux distributions provide binary packages. For scripting languages, the package may be named like 'openbabel-perl' or 'python-openbabel'
Alternatively, with Conda:
conda install openbabel -c conda-forgeAlso available as a snap package:
snap install openbabel
Usage: